
Derivation, validation, and potential treatment implications of novel clinical phenotypes for sepsis.
Seymour CW, Kennedy JN, Wang S, et al.
JAMA. 2019;321(20):2003-2017. DOI : 10.1001/jama.2019.5791.
Question évaluée
En appliquant des méthodes d’intelligence artificielle sur big data 1) Identifier de nouveaux phénotypes de sepsis 2) Déterminer si ces phénotypes sont associés à un profil de biomarqueur ou à un devenir différent 3) Prédire si la distribution de ces phénotypes aurait pu influer sur les résultats d’essais cliniques.
Type d’étude
Analyse rétrospective
Population étudiée
Utilisation d’une population issue de 3 cohortes (SENECA 2010-2012, SENECA 2013-2014, GenIMS) et 3 essais cliniques (ProCESS, ACESS, PROWESS). Patients adultes admis pour sepsis défini selon les critères Sepsis-2 (GenIMS, Essais cliniques) ou Sepsis-3 (2 cohortes SENECA). Le sepsis était diagnostiqué au maximum 6 heures après l’admission à l’hôpital.
Méthode
Etape 1 : Identification des profils
Les auteurs ont utilisé une techniques d’apprentissage non supervisée : l’algorithme des k moyennes. Cette technique est non supervisée car le nombre et la nature des classes ou « clusters » n’est pas déterminée à priori.
Une fois le nombre de profils identifiés, la répartition des individus s’effectue par minimisation de la somme des carrés de la distance entre le point individuel et la moyenne des points du cluster (Figure 1).
Figure 1 : Schéma conceptuel de la clusterisation par algorithme des k moyennes
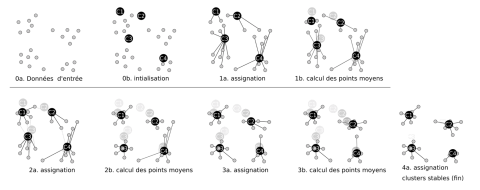
Une analyse de sensibilité par application d’une autre méthodologie (régression par classe latente) a été appliquée pour confirmer le nombre et la nature des profils.
Etape 2 : Evaluer la corrélation entre les phénotypes identifiés
Après identifications des profils, ces derniers ont pu être attribués aux individus des autres cohortes et essais cliniques.
Des analyses « classiques » de comparaison entre les profils identifiés ont été réalisées afin d’évaluer la distribution des biomarqueurs de réponse immunitaire et la survie des patients.
Etape 3 : Prédire l’effet traitement en fonction des phénotypes identifiés
Enfin, l’effet traitement et l’impact des profils sur l’effet traitement a pu être évalué en 2 temps :
- Une étude de simulation par modification de la proportion relative des profils au sein de la population globale.
- Par application d’un modèle de régression logistique en incluant dans le modèle final la variable traitement et le profil.
Résultats essentiels
Quatre profils ont pu être identifiés :
- Profil α: Peu d’anomalie biologique et de dysfonction d’organe. Mortalité faible à 2%.
- Profil β: Patients plus âgés et plus comorbides. Dysfonction rénale. Mortalité à 5%
- Profil γ: Inflammatoire (biomaqueurs inflammatoires élevés, température plus élevée). Mortalité à 15%
- Profil δ: Lactatémie plus élevée, cytolyse hépatique plus importante et hypotension plus marquée. Mortalité à 32%.
Concernant les marqueurs inflammatoires, les profils γ et δ étaient volontiers plus inflammatoires avec une atteinte endothéliale plus sévère et une coagulopathie plus marquées que les 2 autres.
A propos de l’effet traitement, il n’a pas été retrouvé de modification des effets rapportés après analyse ad hoc des sous population de profils. Cependant, les études de simulation d’enrichissement, qui modifiaient peu ou pas les caractéristiques initiales de la cohorte globale, impactaient les effets traitements. Par exemple, au sein de l’étude ProCESS dans laquelle la publication originale ne retrouvait pas d’effet traitement de la stratégie « early goal directed therapy », l’incrémentation du profil α s’accompagnait d’un bénéfice observé tandis qu’une surreprésentation d’un profil δ se traduisait par une nocivité importante de la procédure.
Commentaires
L’Homme a initialement développé le processus de catégorisation à visée de simplification, cette envie de classifier a par la suite évolué vers l’ambition de classer pour mieux comprendre puis pour prédire.
Ce procédé de « clustering » est d’ailleurs un sujet de recherche en intelligence artificielle. Pour les moins à l’aise avec cette méthodologie, on distingue la classification supervisé et non supervisé. Dans le 1ercas, il s’agit d’apprendre à classer un nouvel individu parmi un ensemble de classes prédéfinies. Les classes sont connues à l’avance, c’est ce que nous utilisons dans les processus d’apprentissage. Issu des statistiques, la classification non supervisée comme son nom l’indique, consiste à apprendre sans superviseur. A partir d’une population, il s’agit d’extraire des classes ou groupes d’individus présentant des caractéristiques communes, le nombre et la définition des classes n’étant pas donnée a priori.
S’il est vrai que les définitions du sepsis ont largement évolué vers la simplification, abolissant ainsi les concepts de SIRS et de sepsis sévère, permettant son identification plus précocement, c’est d’un tout autre constat maintenant (« il n’y a pas un sepsis mais des sepsis ») que les auteurs sont partis pour classer et prédire l’évolution des malades. Et l’originalité ici est dans l’établissement d’une classification non pas par porte d’entrée infectieuse, par documentation bactériologique ou par scores de gravité mais par l’utilisation de toutes ces données via l’application des techniques d’IA.
Par ailleurs, les auteurs ont mis en exergue l’instabilité de résultats d’essais cliniques bien connus. Est-ce que pour autant suffisant pour relancer les essais cliniques sur la protéine C activé, l’albumine? Il y a là de quoi enfoncer des portes quasi fermées…
Mais qui dit médecine personnalisée dit meilleure compréhension des processus physiopathologiques sous-jacent. Et si la science des données peut nous orienter vers la ou les directions que prendront les malades, elle n’a de sens qu’associée avec une meilleure compréhension des mécanismes biologiques mis en cause et potentiellement influençables.
Enfin, la qualité de ces études réside en grande partie dans la qualité des données. Et s’il est aisé de récupérer des valeurs biologiques à l’admission des malades, il s’agit d’une toute autre affaire concernant un antécédent, une iatrogénie récente ou tout autre processus impactant l’installation du sepsis.
Si l’idée d’une application quotidienne au lit des malades reste malgré tout très hâtive, l’idée de l’application future des techniques d’IA dans la prédiction et la prise en charge des malades semblent être une perspective à portée de main (3).
Points forts
- Caractère big data.
- Utilisation de techniques de machine learning bien répandues et validées.
- Résultats identifiés pragmatiques et vérifient bien ce que les cliniciens observent au quotidien dans les services.
Points faibles
- Plusieurs étapes clés soumises à la subjectivité des auteurs : choix des variables candidates pour l’identification des profils, normalisation de la distribution de certaines variables imposée par la méthodologie des k moyennes, fenêtre horaire des 6h entre l’admission et le recueil des variables.
- Non prise en compte des processus physiopathologiques sous-jacents.
- Non prise en compte de l’anamnèse récente des malades (iatrogénicité, hospitalisation précédente) pouvant contribuer à l’installation du tableau.
- Utilisation d’une base de donnée issue d’un unique système de base de donnée (SENECA project, University of Pittsburg Medical Center).
CONFLIT D'INTÉRÊTS
Article commenté par Matthieu Jamme, Réanimation polyvalente, CHI Poissy Saint Germain en Laye. Centre de recherche en épidemiologie et santé des populations (CESP), Equipe Rein et Cœur, INSER U1018, Université Paris Sud, Villejuif.
L’auteur déclare ne pas avoir de liens d’intérêt en rapport avec cette REACTU.
Le contenu des fiches REACTU traduit la position de leurs auteurs, mais n’engage ni la CERC ni la SRLF.
Envoyez vos commentaires/réactions à l’auteur (mjamme@ch-poissy-st-germain.fr) ou à la CERC.
LIENS UTILES
- Seymour CW, Kennedy JN, Wang S, et al. Derivation, validation, and potential treatment implications of novel clinical phenotypes for sepsis. 2019. doi :10.1001/jama.2019.5791
- Seymour CW, Liu VX, Iwashyna TJ, et al. Assessment of clinical criteria for sepsis: for the third international consensus definitions for sepsis and septic shock (Sepsis-3). JAMA. 2016;315(8): 762-774. doi: 10.1001/jama.2016.0288.
- Komorowski M, Celi LA, Badawi O, et al. The Articifical Intelligence Clinicians learns optimal treatment strategies for sepsis in intensive care. Nat Med. 2018 ;24(11)1716-1720.doi: 10.1038/s41591-018-0213-5.
CERC
JB. LASCARROU (Secrétaire)
K. BACHOUMAS
SD. BARBAR
G. DECORMEILLE
N. HEMING
B. HERMANN
G. JACQ
T. KAMEL
JF. LLITJOS
L. OUANES-BESBES
G. PITON
L. POIROUX